ARN non codant, le cas du ribozyme LC
Toutes les fonctions du corps sont inscrites dans les gènes d’un individu. Il y a même des morceaux de gènes, des « introns » qui ne codent rien, a priori. Ceux-ci sont normalement coupés au moment de l'étape de transcription, mais pas toujours. La collaboration de plusieurs équipes européennes a permis de mieux comprendre les mécanismes qui permettent à certains introns de réagir activement à l’étape dite « d’épissage », et de réguler la traduction.
Avant d'être traduits en protéines par les ribosomes, les gènes des organismes vivants doivent être transcrits en ARN messager. La séquence codante des messagers doit être continue pour être lue correctement par les particules productrices de protéines que sont les ribosomes. Or les transcrits primaires présentent une alternance de séquences codant des fragments protéiques (exons) et de séquences n'en codant pas (introns). Ces introns doivent être éliminés suivant un processus d'épissage dont les différents mécanismes reposent sur des ARN catalytiques ou ribozymes.
Principalement chez les levures, les champignons et les plantes, des séquences d'introns catalytiques sont intégrées aux transcrits primaires. Ces introns sont considérés comme des séquences parasites car ils peuvent se disséminer dans les génomes et se transmettre entre espèces par des mécanismes propres. Ils véhiculent souvent avec eux des séquences codant pour des protéines de maturation qui facilitent leur dissémination. Les scientifiques ont longtemps cru que la présence de ces introns dans les transcrits primaires était neutre pour l'organisme hôte. Mais de nombreuses observations montrent que ce n'est pas le cas.
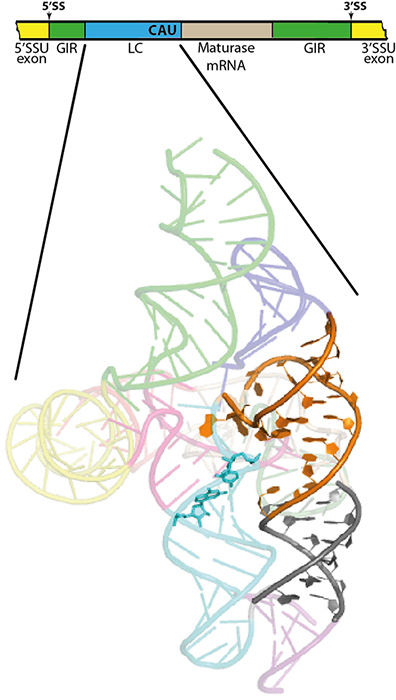
C'est dans ce contexte que les découvertes d’une équipe européenne prennent place. Une partie de l’équipe a d’abord identifié des introns auto-catalytiques qui sont accompagnés d'un ribozyme additionnel qui forme une petite boucle de trois nucléotides à l'extrémité du messager de la maturase codée par l'intron. Cette coiffe en lasso (d'où le nom du ribozyme LC) protège le messager de la maturase de la dégradation et confère donc un avantage sélectif à l'intron en augmentant ses chances de dissémination. Le ribozyme LC, a jusqu'à présent toujours été trouvé dans un transcrit primaire d'ARN ribosomique. Les activités des deux ribozymes doivent donc être coordonnées pour éviter la coupure intempestive du transcrit primaire qui empêcherait la formation des ribosomes et donc la traduction.
Benoit Masquida, co-auteur de l’étude et chercheur dans l'unité Génétique Moléculaire Génomique Microbiologie de Strasbourg (Université Strasbourg, CNRS) détaille les expériences menées sur deux synchrotrons : « les structures cristallographiques et aux petits angles résolues grâce aux données obtenues au Paul Scherrer Institute-Swiss Light Source (x06da) et au synchrotron SOLEIL (lignes PROXIMA 1 et SWING) nous ont permis de mieux comprendre le mécanisme qui permet l'activation du ribozyme LC ainsi que son mécanisme catalytique. De plus, nous avons pu établir que le ribozyme LC dérive d'un intron auto-catalytique qui se serait inséré au sein d'un autre intron où il a subi un processus de spéciation générant des structures spécialisées dans le contrôle d'une nouvelle activité catalytique. »