Altération des propriétés élastiques de la capside du virus de l’hépatite B par des antiviraux
Afin d’enrayer la prolifération de virus pathogènes, une des stratégies consiste à perturber leur assemblage quand ils se multiplient dans les cellules infectées, et ce notamment avec de petites molécules : les modulateurs d’assemblage. Des modulateurs ont été développés pour cibler et déformer la capside du virus de l’hépatite B qui n’est ainsi plus viable pour propager le virus. Les mécanismes physiques d’action de ces antiviraux restent toutefois mal compris.
Un consortium international a montré que les déformations de la capside provoquées par ces modulateurs s’expliquent par une altération des propriétés élastiques des composants de cette capside. La diffusion des rayons X résolue en temps sur la ligne SWING a notamment permis la mesure des énergies d’interaction induites par les modulateurs entre les protéines de la capside.
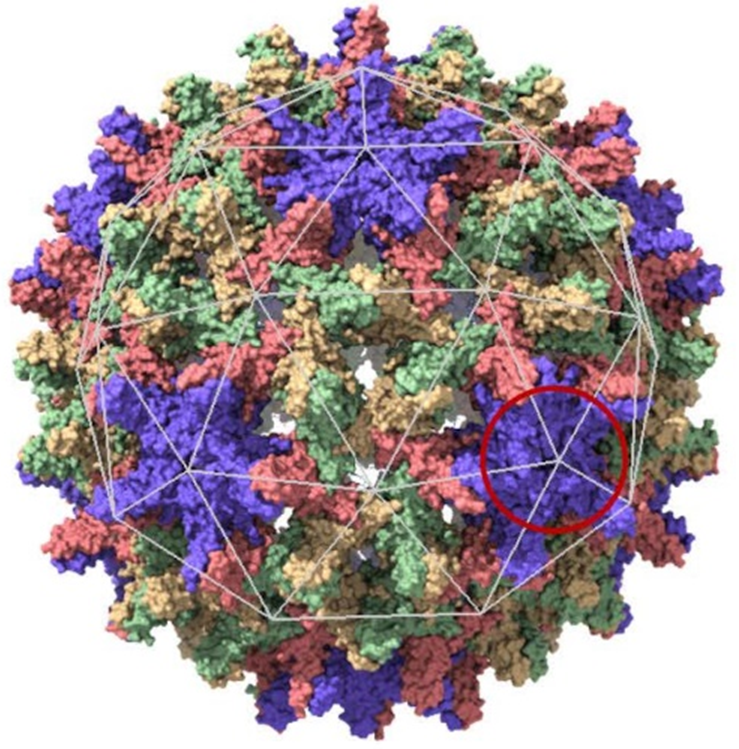
Plus de 250 millions d’individus sont infectés chroniquement par le virus de l’hépatite B (VHB), principalement en Afrique et en Asie du Sud, faute de vaccination systématique. VHB est à l’origine de pathologies sévères telles que la cirrhose du foie ou le carcinome hépatocellulaire conduisant chaque année à la mort de presque un million de patients. VHB est un virus enveloppé comprenant une coque de protéines – la capside – arrangée en une structure de symétrie icosaédrique (structure à 20 faces, Figure 1), protégeant le génome sous forme d’ADN. Lors d’un cycle de vie du virus, les sous-unités – une sous-unité étant un dimère de la protéine de capside – synthétisées par la machinerie de la cellule infectée s’auto-assemblent en capsides avant de franchir la membrane plasmique pour infecter les cellules voisines.
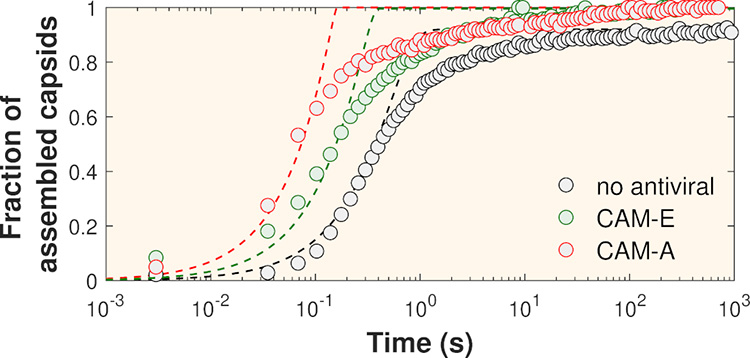
Des chercheurs de l’Université Paris-Saclay ont reproduit in vitro le processus d’auto-assemblage de la capside du VHB en présence de deux types de modulateurs d’assemblage dénommés CAM-E et CAM-A (CAM pour Capsid Assembly Modulator). Lorsque les protéines de capside virale purifiées, initialement conservées dans une solution à faible force ionique, sont brutalement placées dans une solution avec une force ionique physiologique d’environ 150 mM, elles s’assemblent spontanément pour aboutir à un mélange de capsides et de protéines non assemblées. Grâce à la technique de diffusion des rayons X aux petits angles résolue en temps (TR-SAXS) conduite sur la ligne SWING, les chercheurs ont suivi la cinétique d’auto-assemblage avec une haute résolution spatio-temporelle. La Figure 2 présente les fractions massiques de capsides assemblées en fonction du temps déduites des mesures par TR-SAXS. En utilisant un modèle cinétique de transition de phase, les énergies d’interaction entre sous-unités se sont révélées passer de 9kBT sans antiviraux – kBT étant l’énergie thermique – à 18kBT en présence de CAM-A, ce qui signifie que l’interaction entre les protéines formant la capside deviennent deux fois plus fortes en présence de cet antiviral.
Les objets finaux ont été imagés par cryomicroscopie électronique en transmission (cryoTEM) et se sont avérés être soit légèrement ellipsoïdaux en présence de CAM-E, soit très fortement déformés et de plus grande taille avec CAM-A qu’en l’absence d’antiviraux (Figure 3).
Enfin, une équipe de l’Université de Californie à Riverside a simulé l’assemblage de capsides en utilisant un ensemble de triangles (voir Figure 1) et en faisant varier le rapport entre l’énergie d’étirement et l’énergie de courbure des capsides (nombre de Föppl-von Kármán). Ils ont obtenu ainsi des morphologies très similaires aux capsides imagées par cryoTEM (Figure 3).
Cette étude révèle que les modulateurs renforcent considérablement les interactions attractives entre les sous-unités de la capside de VHB, très certainement en augmentant la surface hydrophobe de contact. Des éléments non présentés ici suggèrent de plus que les modulateurs comme CAM-A abaissent l’énergie de courbure de la capside, probablement du fait de leur encombrement stérique qui confère de la flexibilité dans l’angle formé par les sous-unités adjacentes. Hormis la meilleure compréhension des mécanismes d’action des antiviraux, ces résultats pourraient ouvrir des perspectives sur la conception de nanocapsules dérivées de virus avec des morphologies contrôlables.