Régulation cellulaire. Mise en évidence d’une nouvelle cible thérapeutique potentielle, l’ubiquitine-ligase E3 VHL
L'ubiquitinylation des protéines joue, avec la phosphorylation, l'un des rôles les plus importants en régulation cellulaire. Des défaillances dans ces deux voies de signalisation sont impliquées dans les cancers et autres mécanismes pathologiques. Les résultats décrits ici, fruit d’une collaboration entre des chercheurs des universités de Cambridge et Yale, montrent pour la première fois qu’une enzyme intervenant dans l’ubiquitinylation des protéines peut constituer une cible thérapeutique.
L'ubiquitinylation1 des protéines suit une cascade de réactions enzymatiques mettant en jeu une enzyme d'activation de l'ubiquitine (E1), une enzyme de conjugaison de l'ubiquitine (E2) et une ligase de l'ubiquitine (E3). Les ubiquitine-ligases E3 confèrent la spécificité envers le substrat aux voies d'ubiquitinylation des protéines, et constituent donc des cibles thérapeutiques attractives. Toutefois, jusqu'à présent, les efforts pour cibler les ubiquitine-ligases E3 avec de petites molécules ont eu peu de résultats, ce qui a contribué à faire considérer cette famille de protéines comme des cibles « impossibles ».
L’interaction VHL/facteur de transcription Hif-1α, impliquée dans plusieurs pathologies
Les résultats décrits ici montrent pour la première fois que l'ubiquitine-ligase E3 de von Hippel-Lindau (VHL) peut constituer une cible thérapeutique. Ce complexe E3 à sous-unités multiples, composé des protéines pVHL, ÉlongineB, ÉlongineC, Culline2 et Rbx1, joue un rôle majeur dans la détection de l'oxygène par les cellules en catalysant la poly-ubiquitinylation suivie par la dégradation du facteur de transcription Hypoxia Inducible Factor 1 (Hif-1α). Les mutations de pVHL qui affaiblissent ou entravent l'interaction pVHL:Hif-1α, ou qui perturbent l'ubiquitinylation du Hif-1α, conduisent à la maladie de VHL, un syndrome cancéreux humain héréditaire avec une forte occurrence d'hémangioblastomes2 de la rétine, de la moelle épinière et du cervelet, ainsi que de carcinomes à cellules claires du rein et de phéochromocytomes3. Ceci indique une corrélation entre l’activation du Hif-1α et le développement de tumeurs, ainsi qu'un rôle de la ligase E3 de VHL comme suppresseur de tumeur (1).
Mais l'activation de ce facteur Hif-1α possède, d'un autre côté, un impact positif sur des pathologies telles que l'anémie chronique ou les attaques cardiaques. Il s'agit donc d'une protéine-clé, dont l'activation ou au contraire la dégradation joue un rôle dans différentes pathologies.
Plusieurs méthodes pour concevoir des inhibiteurs
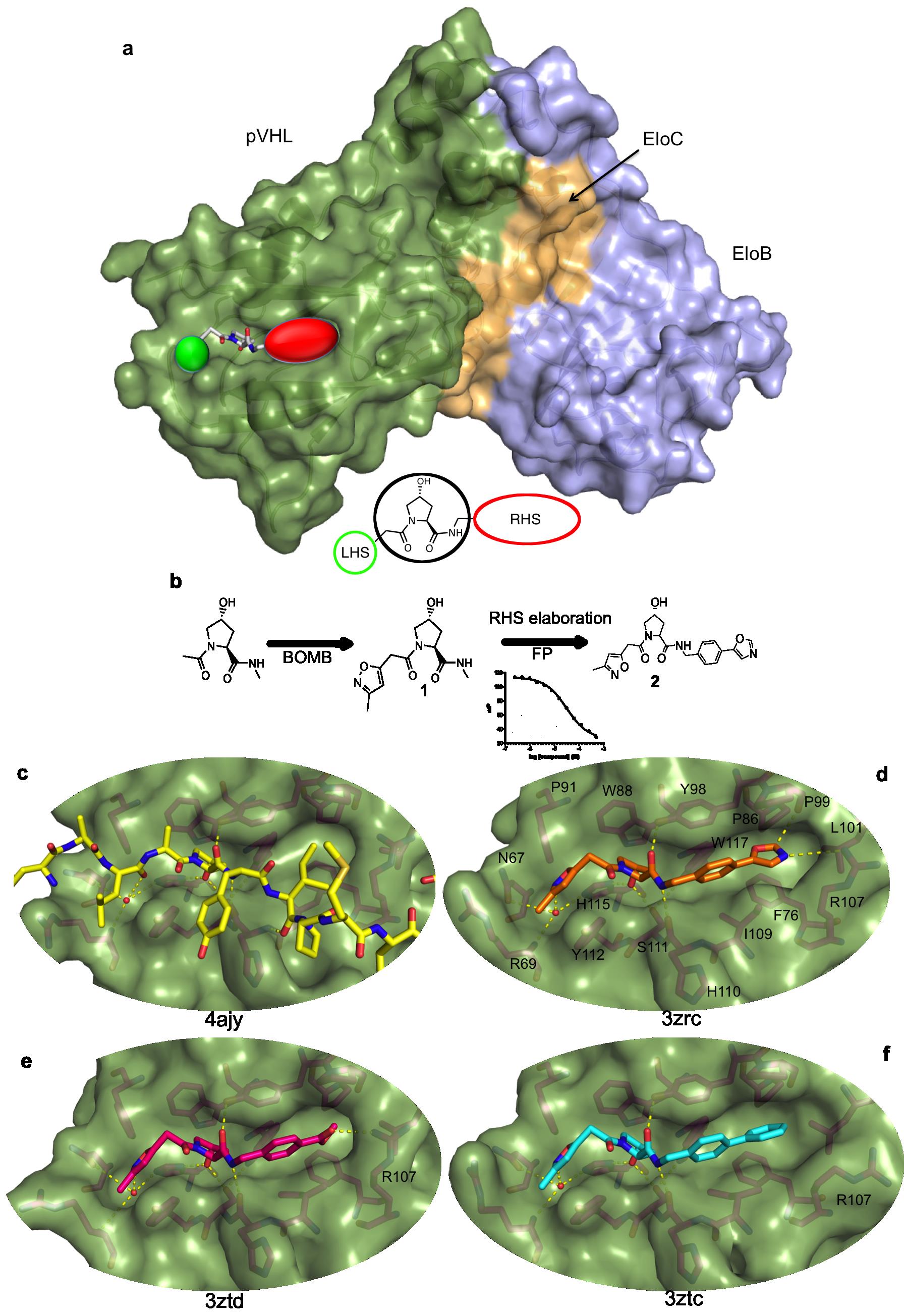
Dans la réf.2 est décrite la découverte des premiers inhibiteurs à affinité micromolaire pour l'interaction pVHL:Hif-1α. Ces composés ont été conçus de manière rationnelle en utilisant comme point de départ le résidu-clé hydroxyproline (Hyp) de Hif-1α qui est crucial pour la liaison entre Hif-1α et pVHL (3-5). Des molécules susceptibles de se lier à ce site constituent des ligands particulièrement intéressants : ils pourraient par la suite cibler des protéines que l'on souhaite détruire par ubiquitinylation, en étant utilisés sous forme de chimère (une partie site de liaison à VHL, une partie site de liaison à la protéine cible à détruire). Grâce à de nouveaux outils de calcul in silico, ont été conçus des analogues de Hyp élaborés pour occuper le côté gauche (LHS) et droit (RHS)de l'interaction protéine-protéine (IPP) entre Hif-1α et pVHL (Figures 1a et 1b). Le composé 1, portant un groupe isoxazole sur le LHS, s'est révélé être un pharmacophore4 prometteur pour interagir avec une molécule d'eau conservée. Des études supplémentaires sur le RHS, à la lumière de nos structures cristallines du pVHL avec les ligands hydratés liés, ont conduit à l'optimisation du composé 2 qui présente des affinités de l'ordre du micromolaire à un chiffre, déterminées par calorimétrie à titrage isotherme (ITC) et polarisation de fluorescence (Figure 1b).
Les structures résolues par diffraction des RX du pVHL avec le composé 2 lié, ainsi qu'un méthylbenzoate et un analogue du biphényle RHS, montrent que les ligands miment bien le mode de liaison du peptide Hif-1α (Figures 1c-f). Ils récapitulent le réseau de liaisons hydrogène autour du résidu central Hyp et forment une liaison hydrogène avec la molécule d'eau dans la poche LHS, conformément à l'hypothèse. D'autre part, les structures cristallines indiquent un certain degré de plasticité conformationnelle de la poche RHS, avec le groupe biphényle de plus grande taille qui induit un mouvement significatif de la chaîne latérale de l’acide aminé Arg107 (Figure 1f) et le groupe oxazole optimal formant des liaisons hydrogène avec le NH2 de Arg107 et avec le carbonyle de la chaîne principale du résidu Pro99 (Figure 1d).
Limites des méthodes classiques de criblage
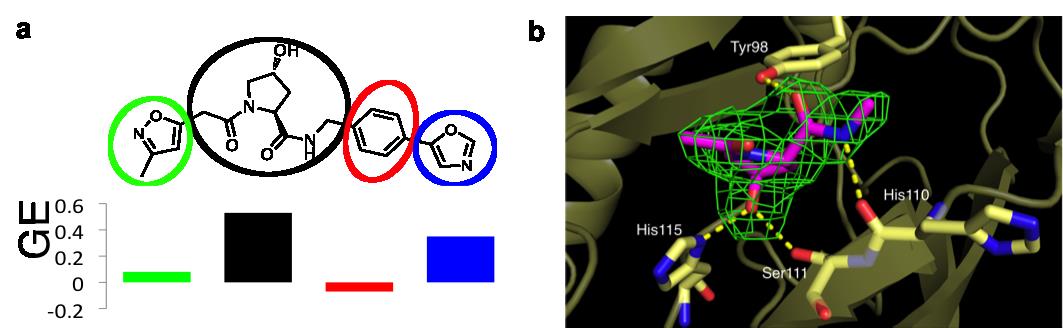
Cette découverte a conduit les auteurs à se demander si ces composés auraient pu être identifiés par une approche basée sur le criblage de fragments. En partant des trois ligands présentés dans la Figure 1, ils les ont « décomposés » de façon modulaire en fragments de plus en plus petits susceptibles de cibler le LHS, le noyau Hyp et le RHS sur le site d'interaction. Les résultats de cette étude sont décrits dans la réf. 6. Avec des techniques biophysiques couramment employées pour le criblage de fragments, les chercheurs ont seulement réussi à détecter la liaison de fragments plus élaborés que les spécimens généralement criblés sur des cibles thérapeutiques « possibles », et qui violent plusieurs directives largement adoptées pour constituer des banques de fragments. En outre, les contributions individuelles à l'énergie libre de liaison du ligand ont été décomposées pour différents groupements, en appliquant des critères de mesure « d'efficacité » qui prennent en compte les affinités de liaison, la taille et la lipophilie. Malgré son affinité modeste qui avait échappé aux moyens de détection dans le criblage biophysique, il apparait que le noyau Hyp est le plus grand contributeur à l'énergie libre de liaison du ligand (Figure 2a) ainsi qu'à la récapitulation des interactions attendues observées dans une structure cristalline du fragment Hyp lié au pVHL (Figure 2b).
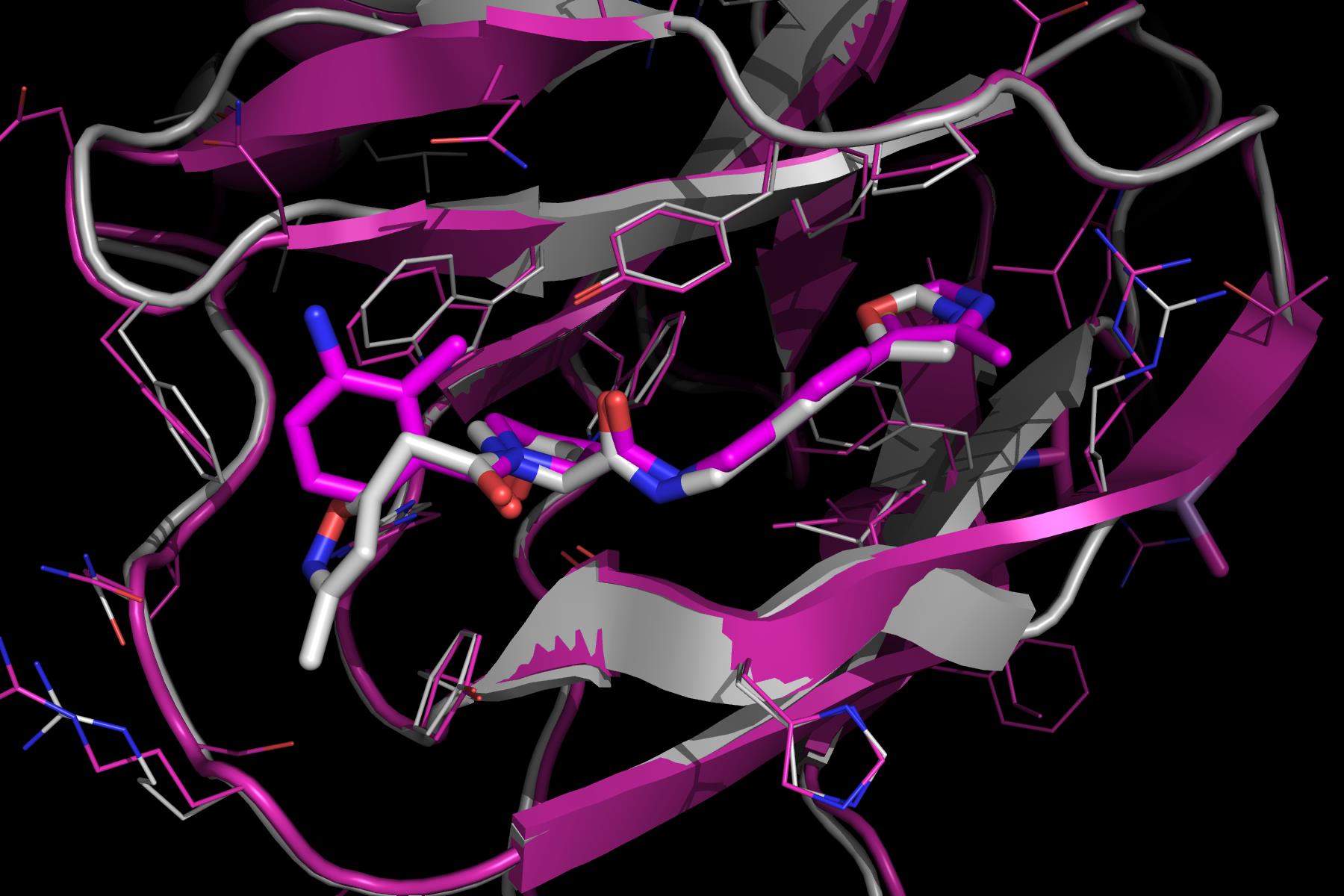
Dans une étude suivante, décrite dans la réf.7, des efforts supplémentaires en chimie pharmaceutique sur les groupes LHS et RHS ont conduit au développement des premiers inhibiteurs sub-micromolaires. Dans un premier stade, le groupe caractéristique oxazole RHS du composé 2 a été optimisé en 4-méthylthiazole. Par la suite, le groupe caractéristique LHS a également été optimisé, ce qui a finalement conduit à l'incorporation d'un groupe 3-amino-2-méthylbenzamide et à un Kd de 0,9 µM. La structure cristalline du composé final (3) montre que le méthylthiazol RHS se lie dans une conformation semblable au groupe oxazole du composé 2 (Figure 3). Il est intéressant de noter que le groupe LHS se lie clairement à pVHL d'une manière alternative. Alors que le groupe isoxazole du composé 2 a interagi avec l'eau structurale comme indiqué ci-dessus (Figure 1d), le benzamide est orienté hors de la poche d'eau et se trouve adjacent à la chaîne latérale du Trp88. L'aniline semble former une liaison hydrogène complètement nouvelle médiée par l'eau avec la chaîne latérale du Gln96.
Conclusion
Ces efforts de conception basée sur la structure 3D ont conduit à la découverte de la première classe efficace de petites molécules ciblant l'ubiquitine-ligase E3 de VHL et affaiblissant son interaction avec le Hif-1α, ce qui prouve la possibilité de cibler cette interaction complexe entre les deux protéines. Une telle approche intégrée combinant la biologie structurale et la chimie pharmaceutique pourrait se révéler utile pour cibler d'autres IPP.
Enfin, et peut-être de manière plus importante, ces résultats mettent en garde les « chasseurs de médicament », cherchant à cibler des IPP à l'aide d'approches basées sur des fragments, sur le fait que d'excellents points de départ chimiques (tels que Hyp) peuvent facilement être négligés ou ratés par les criblages standard. En effet, ils peuvent intervenir dans des liaisons trop faibles pour être détectées par les méthodes conventionnelles, et/ou les fragments peuvent être exclus des banques de fragments conventionnelles.
1 – ubiquitinylation : l'ubiquitine est une petite protéine présente dans tous les compartiments subcellulaires de toutes les cellules des organismes eucaryotes (d’où son nom : elle est ubiquitaire). Elle a pour rôle de marquer d'autres protéines – cette action est l’ubiquitinylation - en vue de leur élimination par protéolyse. Pour cela, plusieurs molécules d'ubiquitine sont liées de façon covalente à la protéine-cible grâce à l'action de trois enzymes, E1, E2 et E3-ligases.
2- hémangioblastome : tumeur vasculaire du système nerveux.
3 - phéochromocytomes : tumeur de certaines cellules de la médullosurrénale (partie centrale des glandes surrénales).
4 – pharmacophore : partie d’une molécule qui est déterminante pour l'activité biologique de cette molécule – à rapprocher du « chromophore » qui est le groupement chimique responsable de la propriété colorée ou colorante d'une molécule.
References
(1) Kaelin, W.G. Nat. Rev. Cancer 2002 2, 673
(2) Buckley, D.L. et al. J. Am. Chem. Soc. 2012 134, 4465–4468
(3) Hon, W-C. et al. Nature 2002 417, 975
(4) Min, J-H. et al. Science 2002 296, 1886
(5) Loenarz, C. et al. Angew. Chem. Int. Ed. 2009 48, 1784
(6) Van Molle, I. et al. Chemistry & Biology 2012 19, 1300
(7) Buckley, D. L. et al. Angew. Chem. Int. Ed. Engl. 2012 51, 11463