La structure cristallographique de l’enzyme PolD suggère un nouveau paradigme pour la classification des ADN polymérases
Dans toutes les formes de vie cellulaire, les enzymes ADN polymérases (ADNPs) jouent un rôle central dans la réplication, la maintenance et la réparation du génome. En conséquence, les ADNPs ont fait l'objet de recherches intensives depuis des décennies. Grâce à des données récoltées sur les lignes PROXIMA-1 et PROXIMA-2A, des équipes de l’Institut Pasteur et de l’IFREMER ont pu résoudre la structure d’une ADNP d’Archée. Leurs résultats révèlent que, si cette enzyme est atypique au sein de la famille des ADNPs, elle partage par contre des caractéristiques avec des ARN polymérases. Ces résultats sont publiés dans Nature Communications.
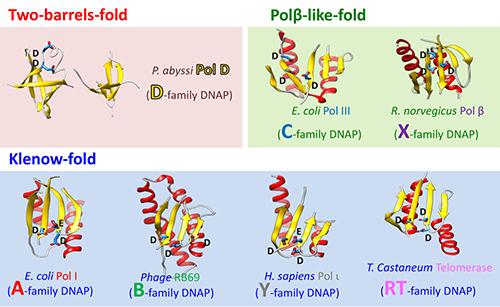
Les ADNPs ont été regroupées en sept différentes familles sur la base d'alignements de leurs séquences en acides aminés : PolA, PolB, PolC, PolD, PolX, PolY et transcriptase inverse (RT). Il est remarquable que la quasi-totalité des ADNPs puissent être regroupées en deux superfamilles structurales: le repliement de type Klenow (PolA, PolB, PolY et RT) et le repliement de type Polβ (PolC et PolX). La dernière famille d'ADNPs pour laquelle la structure n'est pas connue et dont le domaine catalytique n'a pas de repliement associé est la PolD.
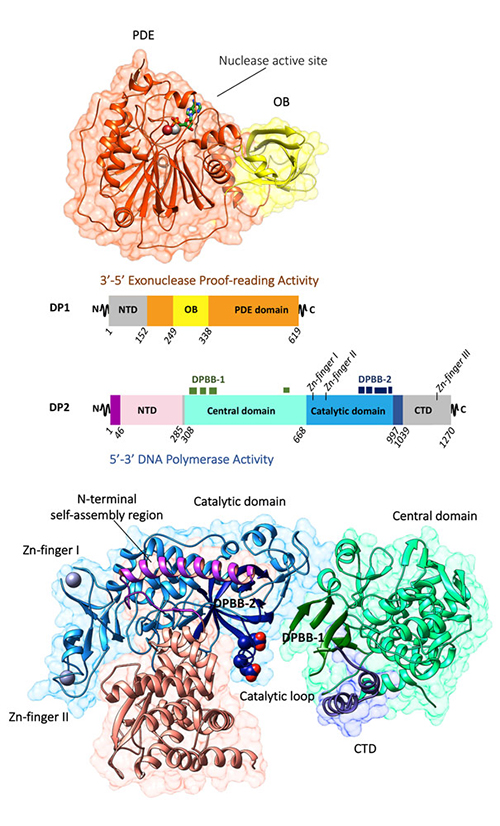
La PolD existe chez tous les Archées*, à l’exception des Crenarchées. C'est une ADNP réplicative responsable de l'initiation de la réplication de l’ADN. Elle est composée d'une grande sous-unité (DP2) portant l'activité 5'-3' polymérase et d'une petite sous-unité (DP1) portant l'activité 3'-5' exonucléase correctrice d'erreurs. Hormis les régions N-terminales des sous-unités DP1 (1-50) et DP2 (50-280), les structures des domaines catalytiques polymérase et exonucléase demeurent inconnues.
Afin de déterminer l'origine évolutive des ADNPs de la famille D, les scientifiques ont résolu la structure cristallographique de deux larges constructions des sous-unités DP1 et DP2 de la PolD de l’Archée Pyrococcus abyssi. Chacune des structures a été déterminée individuellement par phasage expérimental en utilisant la méthode SAD sur les lignes de lumière PROXIMA-1 et PROXIMA-2A, respectivement pour DP2 et DP1 (Figure 1).
Les structures des sous-unités DP1 et DP2 révèlent que la PolD est une ADNP atypique. D’abord, la sous-unité DP1 de relecture exonucléase possède un repliement phosphodiestérase qui est radicalement différent des domaines dnaQ-like retrouvées chez la grande majorité des autres ADNPs. Ensuite, la sous-unité DP2 ne montre aucune similarité structurale avec d'autres ADNPs connues. Avec la structure de DP2, une troisième classe d'ADNPs a été créée (Figure 2).
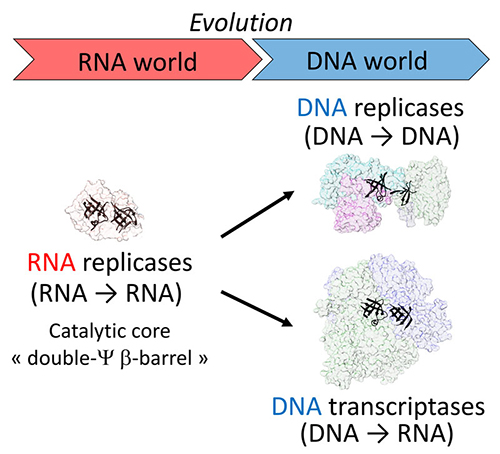
De façon inattendue, les scientifiques ont montré que DP2 partage une homologie structurale avec le repliement en “deux-tonneaux beta” du cœur catalytique des ARN polymérases (ARNPs), incluant les transcriptases multi sous-unités dans les trois domaines du vivant. Cette homologie inattendue définit un nouveau paradigme pour la classification des ADNPs et ARNPs, ainsi qu'une possible origine évolutive commune entre les différents types d'ADNPs et ARNPs. Cela relie pour la première fois, en écartant le monde des virus, la réplication et la transcription de l'ADN au sein d'une même superfamille protéique. Le fait que la PolD et les ARNPs à “deux-tonneaux beta” partagent un cœur catalytique commun suggère que les machineries protéiques responsables de la réplication et de la transcription de l'ADN ont évolué de manière concomitante.
En effet, cette étude suggère que le cœur catalytique à “deux-tonneaux beta” pourrait avoir facilité la transition d’un monde à ARN vers un monde à ADN, en permettant l’évolution conjointe de la réplication et de la transcription à partir d'un cœur catalytique commun, plutôt que les deux types d’enzymes aient aient été « inventées » indépendamment (Figure 3).
* Archées : microorganismes unicellulaires sans noyau, classés depuis quelques décennies dans un groupe différent de celui des bactéries.